© Argaly
Les terres excavées provenant de sites urbains ou industriels potentiellement pollués peuvent être traitées et valorisées. Les sols pollués par métaux lourds sont triés et écartés. Après traitement, les terres peuvent être envoyées en centre de stockage ou préparées pour être valorisées, par exemple en terre végétale par mélange avec différents amendements organiques.
La colonisation de la terre par le vivant a lieu après sa mise en place et dépend de facteurs édaphoclimatiques comme la texture du sol, la nature de l’amendement organique et biotiques (type de couvert végétal, le cas échéant inoculation des sols, etc.).
Dans cette étude, menée par SUEZ Minerals, plusieurs démonstrateurs de sol construit ont été mis en place à grande échelle pour différents usages. Le suivi de ces démonstrateurs consiste en un suivi de la qualité chimique (mobilité des métaux) et agronomique (fertilité) des sols ainsi qu’un suivi de la croissance des végétaux.
SUEZ Minerals a souhaité compléter ce suivi par un indicateur « sol vivant » dont l’objectif est de suivre au cours du temps le retour de la vie du sol (groupes faunistiques, champignons, bactéries) et notamment de comparer la vie ayant repris place dans les sols construits à celle des sols locaux « naturels ».
L’hypothèse de base est que le sol initialement utilisé s’apparente à une matrice minérale quasiment stérile à l’exception des microorganismes. L’ajout de certains types d’amendements organiques (composts, fibres végétales, boues organiques, etc.) et la recréation d’un horizon humifère superficiel (aération, humidification, etc.) permettront la restauration des réseaux trophiques de ces sols. On suppose donc que la colonisation débutera par des décomposeurs de la matière organique apportée (bactéries, champignons), qui seront suivis de consommateurs primaires puis secondaires.
SUEZ Minerals a sollicité ARGALY pour évaluer la possibilité d’établir l’indicateur « sol vivant » à partir de l’ADN extrait des échantillons de sol. ARGALY est une société spécialisée dans les analyses de biodiversité à partir d’ADN environnemental, c’est-à-dire l’ADN extrait à partir d’échantillons environnementaux. La mission d’ARGALY est de proposer des outils innovants et non invasifs pour obtenir des informations taxonomiques ou fonctionnelles dans les écosystèmes terrestres.
Dans le cadre de cette étude, l’ADN du sol de plusieurs démonstrateurs a été extrait, amplifié par PCR pour être ensuite séquencé à l’aide de séquenceurs nouvelle génération. Suite au traitement bio-informatique des données, les organismes présents dans les échantillons de sol ont été identifiés. Différents groupes taxonomiques ont été ciblés afin d’évaluer la biodiversité globale du sol :
- Les eucaryotes : de très nombreux organismes répartis sur toute la chaîne trophique du sol (champignons, lombrics, nématodes, acariens, collemboles, etc.) peuvent être évalués.
- Les bactéries : la diversité de la microflore bactérienne du sol peut ainsi être étudiée, sachant que les fonctions bactériennes sont en général mieux étudiées que les fonctions des eucaryotes.
Cette étude, commencée il y a 2 ans, a permis d’observer qu’à T0, les communautés sont plus proches des communautés identifiées chez les amendements.
D’autre part, les communautés évoluent vers des terres végétales locales matures de manière assez rapide, étant donné que cette évolution est déjà observable après 6 mois. Cette évolution est surtout évidente chez les bactéries, moins chez les eucaryotes, donc le temps de réponse semble différent pour les deux compartiments biologiques.
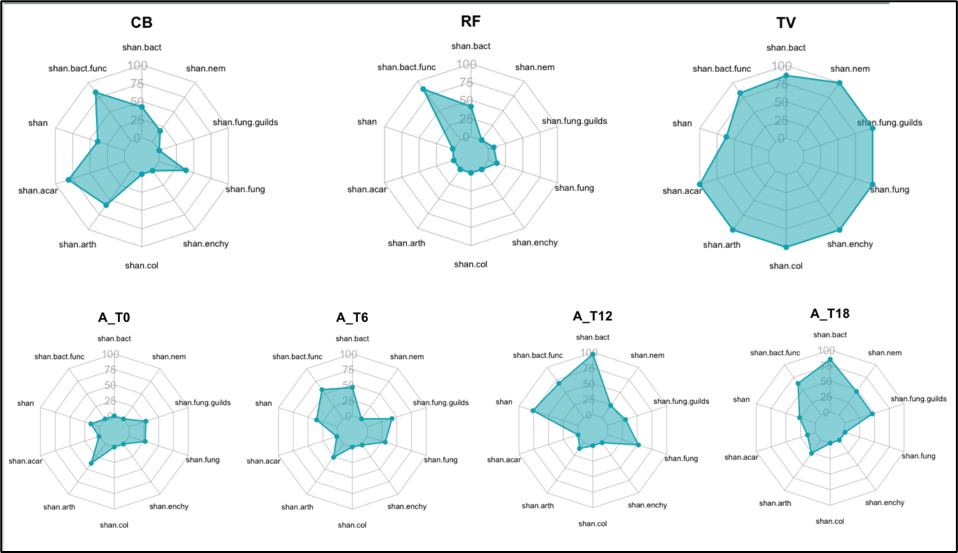
Les diagrammes en radar (exemple pour un démonstrateur en Figure 1) illustrent l’évolution de la diversité des organismes du sol dans le temps.
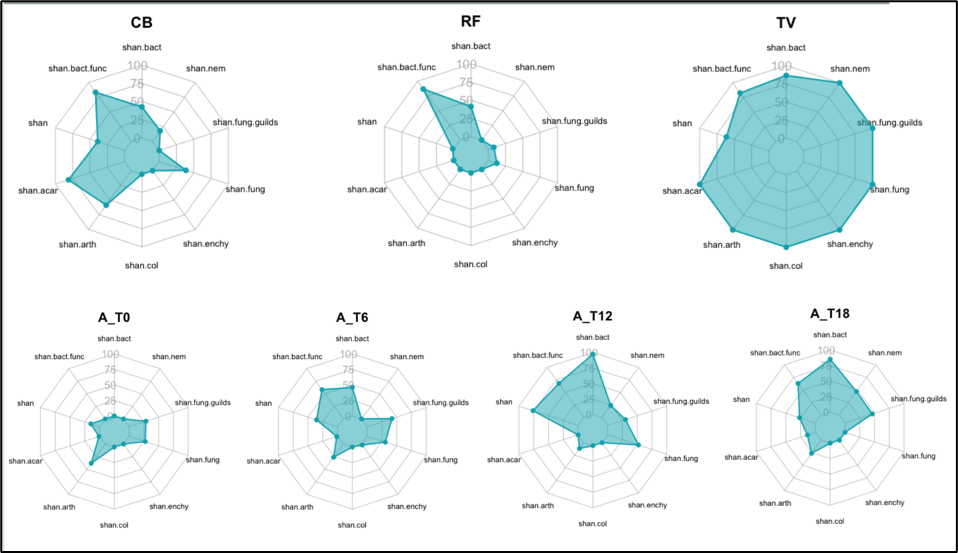
Figure 1. Représentation de la diversité des organismes du sol dans des amendements organiques (partie haute de la figure) et dans des terres excavées et amendées avec un suivi dans le temps. (T0, 6 mois, 12 mois et 18 mois plus tard) (partie basse de la figure). © Argaly
Légende
CB : Compost boue
RF : Refus fibreux
TV : Terre végétale
Shan = Diversité de Shannon
Shan.bact.func : diversité fonctionnelle des bactéries
Shan : Diversité globale
Sahn.acar : Diversité des acariens
Shan.col : Diversité des collemboles
Shan.enchy : Diversité des enchytréides
Shan.fung : Diversité des champignons
Shan.fung.guilds : Diversité des guildes de champignons