Contexte et objectifs
Actuellement, la propagation de la renouée du Japon, une plante exotique envahissante connue pour ses capacités de dispersion et de résistance hors normes suscite de vives inquiétudes. Cette plante pose des défis considérables pour les projets de gestion du territoire et la protection de la biodiversité. Son élimination est possible mais très couteuse avec les techniques connues actuelles. Il est par conséquent essentiel de pouvoir la détecter précocement. Dans ce contexte, Argaly se présente avec une mission claire : détecter cette espèce dans l’eau grâce à des analyses d’ADN environnemental. Avec cet outil innovant, la gestion de cette espèce le long des cours d’eau pourrait devenir plus efficace et proactive, contribuant ainsi à maintenir l’équilibre des écosystèmes et les services fournis.
Originaire d’Asie et importée en Europe au XIXe siècle pour l’ornementation, la renouée du Japon est reconnue comme l’une des espèces exotiques envahissantes les plus problématiques à l’échelle mondiale. Elle impacte fortement la biodiversité et aussi certaines activités humaines. Sa dispersion se fait essentiellement par fragmentation et déplacement de ses rhizomes ou ses tiges.
Dans les cours d’eau, ce sont les crues qui facilitent sa dissémination naturelle. Différentes techniques existent aujourd’hui pour gérer cette plante mais nécessitent des ressources humaines et financières considérables.
C’est pourquoi, sa détection précoce est un enjeu essentiel notamment pour les gestionnaires de cours d’eau, qui doivent visiter un chevelu très important de réseau hydrographique.
Face à cette difficulté, Argaly cherche à développer un outil basé sur l’ADN environnemental (ADNe), une méthode qui permet de détecter les traces d’ADN laissés ou relarguées par les organismes dans l’environnement. La renouée étant lessivée par la pluie et ses rhizomes et tiges déplacées lors des crues, l’hypothèse faite est que son ADN pourrait être retrouvé dans les cours d’eau.
L’objectif est donc de montrer qu’une approche ADNe permettrait de détecter la présence de la plante en aval dans le cours d’eau, ce qui faciliterait le travail des gestionnaires.
Une étude préliminaire a donc été menée en collaboration avec la SCOP Aquabio spécialiste de cette plante (Bourget-du-lac, Savoie) dans l’objectif de voir si l’ADNe de l’espèce pouvait être détecté et dans quelles conditions. Les échantillonnages ont eu lieu sur trois rivières. Pour chaque rivière, trois sites ont été choisis en aval d’un plan de renouée. La surface de sol occupée par la plante était variable selon le cours d’eau et les sites d’échantillonnages ont été choisis à différentes distances en aval du plan de renouée pour voir jusqu’à quelle distance l’ADN pouvait être détecté (Figure 1). De plus, deux sessions d’échantillonnages ont été réalisées pour chaque site selon deux conditions météorologiques différentes pour savoir dans quelle situation l’ADN de la plante allait être le plus détecté (Image 1).
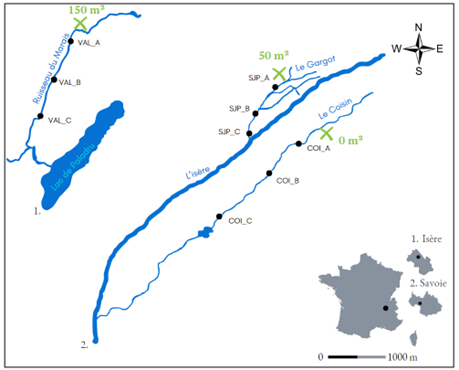


Image 1 : Échantillonnage du site SJP (a) après deux jours de pluie et (b) après deux jours de soleil.
Par la suite, l’ADN des échantillons a été extrait, traité et analysé par Argaly. A cette étape deux méthodes d’ADN environnemental ont été testées afin de comparer leur capacités de de détection dans le cas de la renouée du Japon :
• Le métabarcoding, une approche basée sur des techniques de séquençage haut débit, qui va cibler et séquencer des courts fragments d’ADN taxonomiquement informatifs. Elle est généraliste et communique des informations de présence ou absence sur une communauté ciblée.
• La PCR quantitative, une approche qui va cibler uniquement l’espèce étudiée et va aussi indiquer si l’espèce est présente dans le milieu. Cette dernière est plus rapide et moins coûteuse.
Le graphique de la Figure 2 représente le nombre de lectures de séquençage attribuées à la séquence de la renouée du Japon en fonction de la distance par rapport au plan de renouée, des deux conditions météorologiques et de la taille de la plante. Des résultats similaires ont été trouvés pour la PCR quantitative.
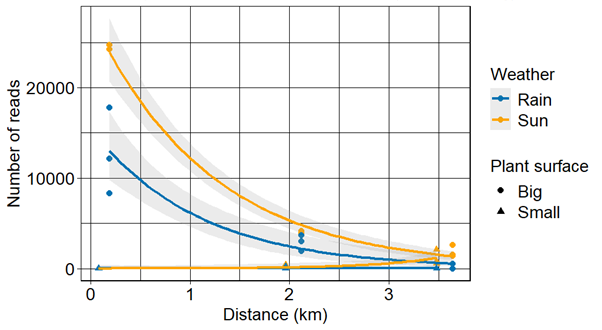
Les résultats de cette étude ont montré que les méthodes d’ADN environnemental pouvaient détecter des traces de renouées dans les cours d’eau. La qPCR et le métabarcoding ont montré des résultats concordants et des capacités de détection quasiment équivalentes. L’ADN de la renouée du japon a été détecté à plus de 3km en aval du patch pour les deux méthodes. De plus, les conditions ensoleillées lors de l’échantillonnage semblent être préférables pour la détection quel que soit la surface occupée par la plante. Enfin la détection semble être plus complexe quand la plante occupe une surface plus faible. D’après ces premiers résultats, l’utilisation de la qPCR comme approche préliminaire permet d’obtenir des informations sur la présence/absence de l’espèce. Cependant l’interprétation des résultats avec cette approche est assez limitée et la détection de faux positifs est compliquée à vérifier. Dans ce cas, un séquençage pour vérifier l’absence d’amplifications non spécifiques pourrait être envisagé.
Cette étude souligne l’efficacité de l’utilisation de l’ADN environnemental pour détecter la présence de la renouée du Japon en amont d’un point d’échantillonnage afin d’alerter le gestionnaire sur sa présence. Cependant, il est important de noter que la détection peut être affectée par divers facteurs environnementaux tels que la distance par rapport au point d’échantillonnage et la taille du cours d’eau. Ainsi, des études complémentaires visant à vérifier la fiabilité de cette approche sont recommandées. Ces études permettraient de fournir aux gestionnaires des informations essentielles pour prendre des décisions éclairées sur le terrain.
Abigaël Chieux, alternante en M2 chez Argaly